Cross Validation¶
Cross Validation의 필요성.¶
- 전체 데이터가 적은 경우, test dataset에 최소한의 데이터를 나누어 주었다고 해도 training dataset에 있는 데이터의 양이 적어질 수 밖에 없음.
- 이같이 적은 데이터에서 hyper parameter tuning을 위한 validation dataset을 다시 나눌 경우, 통계적으로 신뢰할 수 있는 evaluation이 어렵다.
- 지나치게 적은 validation dataset으로는 적절한 model selection이나 hyper parameter tuning이 수행될 수 없게 된다.
Cross Validation (CV) 이란?¶
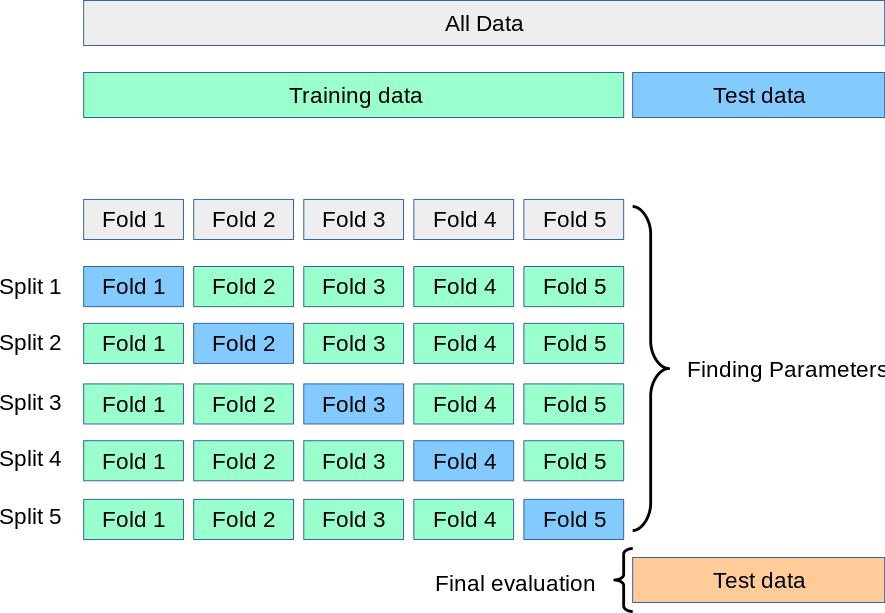
이를 해결하기 위한 방안 중 하나가 k-fold cross validation으로,
- training dataset을
k개의 subset (fold라고도 부름)으로 나누고, - 각 subset을 validation set으로 삼고
- 나머지로 학습을 한 model에 대해 evaluation하는 것을 k번 반복한다.
이 경우, k번의 evaluation의 결과를 얻게 되므로 1번만 수행하는 evaluation에 비해 보다 높은 통계적 신뢰성을 확보할 수 있다.
즉, 하나하나의 evaluation이 적당한 양의 validation dataset을 가지기 어려워 신뢰도가 떨어지니, 이를 여러번 반복 측정하여 신뢰도를 올리는 것이다.
극한의 k-fold CV는 Leave One Out Cross Validation (LOOCV)로
- 1개의 sample만을 validation dataset으로 처리하고
- 해당 1개의 sample을 제외한 나머지로 학습한다.
- 이후 해당 sample로만 evaluation 수행한다.
- 위의 과정을 전체 sample수만큼 반복하는
CV(교차검증)이다.
scikit-learn에서는 다양한 방식의 cross validation을 지원함:
Split 객체 사용하기.¶
scikit-learn에서는 Split 객체 라는 용어 대신 Validation Iterator라고 부름.
train_test_split 함수와 달리,
- split 객체들로 처리하는 방법은
- split 객체의 메서드
split를 통해 실제 training dataset과 validating dataset을 나눔.
- split 객체의 메서드
- 앞서 언급한 cross validation을 보다 세밀하게 제어 가능함.
scikit-learn은 다양한 Split 클래스를 제공함.
sklearn.model_selection.KFold: 순서대로 fold를 나누는 것 주의(shuffle 처리 필요)sklearn.model_selection.StratifiedKFold: target이 각 fold별로 비슷한 비율로 분포sklearn.model_selection.GroupKFold: 그룹이 같은 fold에 놓이도록 처리됨.sklearn.model_selection.ShuffleSplit: 각 모델 훈련 및 검증을 한번 할 때, 랜덤하게 sampling.sklearn.model_selection.StratifiedShuffleSplitsklearn.model_seleciton.RepeatedKFold: KFold를 반복하여 더 높은 통계적 신뢰도를 획득.sklearn.model_selection.RepeatedStratifiedKFoldsklearn.model_selection.LeaveOneOut: LOOCV 수행.sklearn.model_selection.LeavePOut: LOPCV 수행.
sklearn.model_selection.StratifiedKFold¶
K-Fold 로 나눌 때, stratified sampling을 수행 하는 split 클래스.
scikit-learn에서 classification의 경우
cross_val_score및cross_validate에서 기본 Split 객체로 사용됨.
regression의 경우에는
기본으로KFold가 사용됨.
이를 변경하기 위해선cv파라메터에 직접 사용할 Split 객체를 지정하면 됨.
- 이때 stratum을 나누는 기준은 target (or label)임: target class 분포가 각 fold에서 균일하도록 나누어짐.
n_splits에 할당하는 argument로cv에서 사용할number of folds를 지정한다.- 만약
1을 사용하면,train_test_split함수와 같은 역할을 수행하게 됨 (물론 이후split를 호출).
StratifiedKFold는 k-Fold 기법을 구현한 것으로 test dataset으로 지정된 index들이 겹쳐지지 않는다.
사용법은 다음과 같음.
from sklearn.model_selection import StratifiedKFold
kfold = StratifiedKFold(
n_splits, # num of folds
shuffle = False, # index를 나누기 전 랜덤하게 섞을지 여부.
random_state, # seed value for pseudo-random (shuffle이 True인 경우만 설정)
)
for fold_idx, (train_idx, test_idx) in enumerate(kfold.split(X_data, y_data)):
...
sklearn.model_selection.StratifiedShuffleSplit¶
앞서 설명한 StratifiedKFold와 달리 각 반복에서 샘플링을 할 때, shuffling을 하고 train set과 test set을 가하기 때문에,
각 반복에서 test set에 이전 반복의 sample이 중복될 수 있음.
n_splits에 할당하는 argument에 따라 CV에서 이루어질 evaluation이 몇 번 이루어질지를 결정한다.
k-Fold와 달리 램덤하게 test dataset이 결정 되며- test에 선택된 index들이 겹칠 수 있다.
- 10번의 model이 훈련된다고 할 때, 특정 sample point는 2번 이상 test set에 속할 수 있음.
k-Fold에서는 subset이 나누어지는 구조이기 때문에 test dataset이 겹칠 수가 없다- 모든 샘플은 test dataset이 될 수 있는 것은 오직 하나의 fold로만 한정됨.
- 같은 반복에서 하나의 data point 가 여러번 사용될 수는 없고 한 번만 사용됨:
- Bootstrap Sampling에서는 sampling with replacement (복원샘플링) 인 것과 달리,
StratifiedShuffleSplit에서는 sampling without replacement임.- model 의 훈련과 테스트가 수행되는 각 단계에서 data sample은 test set 또는 training set 중 하나에만 속한다는 애기임.
from sklearn.model_selection import StratifiedShuffleSplit
shuffle_splitter = StratifiedShuffleSplit(
n_splits, # num of evaluations
random_state, # seed value for pseudo-random
test_size = 0.3 # k-Fold와 달리 랜덤하게 test dataset을 만들므로 한번의 evaluation에 몇개를 사용할지를 test_size로 결정해줌.
)
for fold_idx, (train_idx, test_idx) in enumerate(shuffle_splitter.split(X_data, y_data)):
...
예제 : index를 통한 차이점 확인¶
StratifiedShuffleSplit과 StratifiedKFold의 차이를 보여주는 예제코드임.
test set 의 index의 분포를 살펴보면 그 차이를 알 수 있음.
import numpy as np
from sklearn.model_selection import StratifiedKFold
np.random.seed(23)
X_data = np.array([ -10, -8, 1 ,2 ,3, 4 , 5, 6, 7, 0])
y_data = np.array([ 0, 0, 1, 1, 1, 1, 1, 1, 1, 0])
n_split = 3
kfold = StratifiedKFold(
n_splits=n_split, # num of folds
# shuffle = False, # index를 나누기 전 랜덤하게 섞을지 여부.
# random_state = 23, # seed value for pseudo-random
)
for fold_idx, (train_idx, test_idx) in enumerate(kfold.split(X_data, y_data)):
print('----------------------')
print(f'fold idx : {fold_idx}')
print(f'train_idx = {train_idx}')
print(f'test_idx = {test_idx}')
결과는 다음과 같다.
----------------------
fold idx : 0
train_idx = [1 5 6 7 8 9]
test_idx = [0 2 3 4]
----------------------
fold idx : 1
train_idx = [0 2 3 4 7 8 9]
test_idx = [1 5 6]
----------------------
fold idx : 2
train_idx = [0 1 2 3 4 5 6]
test_idx = [7 8 9]
각 sample들이 단 한번만 test dataset에서 사용됨을 확인할 수 있다. (각 fold당 겹쳐지지 않는다)
이에 비해 StratifiedShuffleSplit는 test_size로 지정하기 때문에 여러번의 CV가 가능하다.
from sklearn.model_selection import StratifiedShuffleSplit
shuffle_spliter = StratifiedShuffleSplit(
n_splits = n_split, # num of evaluations
random_state = 23, # seed value for pseudo-random
test_size = 0.3 # k-Fold와 달리 랜덤하게 test dataset을 만들므로 한번의 evaluation에 몇개를 사용할지를 test_size로 결정해줌.
)
for shuffle_idx, (train_idx, test_idx) in enumerate(shuffle_spliter.split(X_data, y_data)):
print('----------------------')
print(f'shuffle idx : {shuffle_idx}')
print(f'train_idx = {train_idx}')
print(f'test_idx = {test_idx}')
결과는 다음과 같다.
----------------------
shuffle idx : 0
train_idx = [1 5 7 0 4 6 8]
test_idx = [9 3 2]
----------------------
shuffle idx : 1
train_idx = [6 4 1 5 3 8 9]
test_idx = [7 0 2]
----------------------
shuffle idx : 2
train_idx = [4 1 7 0 2 3 8]
test_idx = [6 5 9]
- 2번과 9번 샘플은 2차례 test set에 속함.
- 하지만 각 shuffle idx 로 한정할 경우, 각 샘플은 test set과 training set 중에 하나에만 속하고, 한번씩만 사용됨.
cross_val_scores()¶
cross_val_scores() 를 사용하면 간단하게 cv 수행 가능.
scores = cross_val_score(
estimator, # CV를 수행할 estimator
X, # Predictors (or feature vectors)
scoring=None, # CV에 사용할 metric. 간단하게는 'accuracy'등이 사용됨.
cv=None, # num of folds.
n_jobs=1, # 사용할 cpu수. -1 일 경우 사용가능한 모든 cpu core를 사용.
verbose=0, # 출력할 메시지의 정도를 결정.
fit_params=None,
pre_dispatch=2*n_jobs
내부적으로
StratifiedKFold를 사용.
좀 더 세밀한 반환값이 필요하다면,cross_val_predict를 사용할 것: predicted value를 반환.
scoring: cross validation에서 사용할 custom function을 지정할 수도 있음.- 일반적으로 utility score가 사용되므로, cost function등이 사용될 경우, negation이 필요함.
- 기본값(None)은 회귀 문제에서
"r2", 분류 문제에서"accuracy"로 설정됨 sklearn.metrics.make_score를 통해 custom score 사용가능(아래 예제코드 참고)- dictionary를 이용하여 복수 개의 scores를 계산하도록 처리 가능함.
fit_params: fit 메소드에 추가로 전달할 파라미터.- 예를 들어, 특정 모델에서 fit 시 추가 옵션이 필요하면 딕셔너리 형태로 전달 가능.
- 기본값은
None.
pre_dispatch='2*n_jobs': 병렬 처리에서 작업을 얼마나 미리 분배할지를 결정.- 기본값은 '2*n_jobs'로, 두 배의 작업량을 미리 준비하여 처리 속도를 높이는 효과.
n_jobs가 클 경우, memory usage가 매우 커질 수도 있으니 유의할 것.
참고: Coefficient of Determination \(r^2\)에 대한 설명
다음의 코드는 LinearRegression을 통해 diabetes dataset에 대해 5fold CV를 수행하는 예제임.
import numpy as np
from sklearn.datasets import load_diabetes
from sklearn.model_selection import cross_val_score, cross_val_predict
from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import make_scorer
# RMSPE 계산 함수 정의 (y_true가 0인 경우 처리)
def rmspe_func(y_true, y_pred):
"""
RMSPE 계산 함수
:param y_true: 실제 값 (numpy array)
:param y_pred: 예측 값 (numpy array)
:return: RMSPE 값 (% 단위)
"""
# y_true가 0인 경우를 처리하기 위한 mask 생성
mask = y_true != 0 # y_true가 0이 아닌 경우에만 계산하도록 마스킹
rmspe_value = np.sqrt(
np.mean(
np.where(
mask, # 마스크가 True인 경우만 계산
((y_true[mask] - y_pred[mask]) / y_true[mask]) ** 2, # RMSPE 계산
0, # y_true가 0인 경우 0으로 처리
)
)
)
return rmspe_value * 100 # RMSPE를 % 단위로 반환
# 1. 데이터 로드
diabetes = load_diabetes() # 당뇨병 데이터셋 로드
X = diabetes.data # 특성 데이터
y = diabetes.target # 타겟 값
# 2. 데이터 표준화
scaler = StandardScaler() # 데이터를 표준화하기 위한 스케일러 정의
X_scaled = scaler.fit_transform(X) # X 데이터를 표준화 (평균 0, 표준편차 1)
# 3. Linear Regression 모델 정의
lr = LinearRegression() # 선형 회귀 모델 정의
# 4. cross_val_score: 교차 검증으로 모델 평가 점수 계산
# 사용자 정의 함수인 RMSPE를 스코어링 함수로 사용
rmspe_scorer = make_scorer(
rmspe_func, # 앞서 정의한 RMSPE 함수를 스코어링 함수로 변환
greater_is_better=False, # 낮을수록 더 좋은 성능으로 평가
)
# cross_val_score를 사용하여 5겹 교차 검증 수행, 각 폴드에 대한 RMSPE 계산
scores = cross_val_score(
lr, # 선형 회귀 모델
X_scaled, # 표준화된 특성 데이터
y, # 타겟 값
cv=5, # 5겹 교차 검증
scoring=rmspe_scorer, # 사용자 정의 RMSPE 스코어링 함수 사용
)
# RMSPE는 음수로 반환되므로 양수로 변환
rmspe = -1. * scores
# 결과 출력 (cross_val_score)
print(f"Cross-validated RMSPE: {np.round(rmspe,4)}") # 각 폴드의 RMSPE 출력
print(f"Mean RMSPE: {np.mean(scores):.4f}") # RMSPE 평균 출력
print(f"Standard deviation of RMSPE: {np.std(scores):.4f}") # RMSPE 표준편차 출력
print('------------------')
# 5. cross_val_predict: 교차 검증을 통한 예측값 반환 (좀 더 다양한 분석을 직접할 수 있음)
y_pred = cross_val_predict(
lr, # 선형 회귀 모델
X_scaled, # 표준화된 특성 데이터
y, # 타겟 값
cv=5 # 5겹 교차 검증
)
# RMSE 계산 (cross_val_predict)
rmspe_cross_val_predict = rmspe_func(y, y_pred) # 교차 검증을 통해 얻은 예측값으로 RMSPE 계산
# 결과 출력 (cross_val_predict)
print(f"{y_pred.shape = }") # 예측값의 형상 출력
print(f"RMSPE with cross_val_predict: {rmspe_cross_val_predict:.4f}") # cross_val_predict를 사용한 RMSPE 출력
결과는 다음과 같음:
Cross-validated RMSPE: [62.8598 71.6347 59.3087 53.9604 61.7267]
Mean RMSPE: -61.8981
Standard deviation of RMSPE: 5.7527
------------------
y_pred.shape = (442,)
RMSPE with cross_val_predict: 62.1894
cross_validate¶
좀 더 자세한 제어를 위해서는 cross_validate 함수를 사용한다.
cross_val_score는 cross_validate의 반환값에서 test_score 키에 해당하는 값만을 반환하는 간단한 버전임.
cross_validate 함수의 반환 객체와 그 활용은 다음과 같음:
- 반환 객체의 타입:
cross_validate함수는dict타입의 객체를 반환.
- 반환 객체의 주요 키:
test_score: 각 fold의 테스트 점수fit_time: 각 fold의 학습 시간score_time: 각 fold의 점수 계산 시간estimator: 각 fold에서 학습된 모델 (return_estimator=True일 때만)
- predict 수행하는 방법.:
cross_validate는dict객체를 반환하므로, 직접 반환되는 객체로predict나score등의 메서드를 호출하는 것은 불가함.- 하지만
return_estimator=True로 설정하면, 각 fold에서 학습된 model들을 얻을 수 있음. - 이 모델들을 사용하여 새로운 데이터에 대한 예측이나 점수 계산을 수행 가능함.
- 또한, 각 fold의 점수나 학습/평가 시간 등의 정보를 통해 모델의 전반적인 성능과 안정성을 평가할 수 있음.
다음의 예제를 참고할 것.
from sklearn.model_selection import cross_validate
from sklearn.datasets import load_iris
from sklearn.svm import SVC
from sklearn.metrics import make_scorer, accuracy_score, precision_score
import numpy as np
# 데이터 로드
iris = load_iris()
X, y = iris.data, iris.target
# 모델 생성
model = SVC(kernel='rbf', random_state=42)
# 사용자 정의 스코어러 생성
precision_scorer = make_scorer(precision_score, average='macro')
# cross_validate 함수 호출 (파라미터 상세 설명)
cv_results = cross_validate(
estimator=model, # 평가할 모델
X=X, # 특성 데이터
y=y, # 타겟 데이터
scoring={'accuracy': 'accuracy', 'precision': precision_scorer}, # 평가 지표
cv=5, # 교차 검증 분할 수
return_train_score=True, # 훈련 세트 점수 반환
return_estimator=True, # 각 분할에 대해 훈련된 추정기 반환
n_jobs=-1, # 모든 가용 CPU 사용
verbose=1, # 진행 상황 출력
error_score='raise' # 오류 발생 시 예외 발생
)
# 반환 객체 정보 출력
print("\n반환 객체의 타입:", type(cv_results))
print("반환 객체의 키:", cv_results.keys())
# 결과 출력
print("\n테스트 정확도:", cv_results['test_accuracy'])
print("테스트 정밀도:", cv_results['test_precision'])
print("평균 테스트 정확도:", cv_results['test_accuracy'].mean())
print("평균 테스트 정밀도:", cv_results['test_precision'].mean())
# 최고 성능 모델 선택 (정확도 기준)
best_model_index = np.argmax(cv_results['test_accuracy'])
best_model = cv_results['estimator'][best_model_index]
# 새 데이터에 대한 예측
new_data = np.array([[5.1, 3.5, 1.4, 0.2]]) # 예시 데이터
prediction = best_model.predict(new_data)
print("\n새 데이터에 대한 예측:", prediction)
# 전체 데이터셋에 대한 점수 계산
score = best_model.score(X, y)
print("전체 데이터셋에 대한 점수:", score)
where,
test_accuracy는 각각의 test set에서의 성적으로cv로 지정한 fold 수 만큼 나옴.train_accuracy는 각각의 train set에서의 성적임.- 앞서 설명한대로
cv파라메터에는Split객체를 넘겨주어 보다 정밀한 제어가 가능함.
참고: Bootstrap Sampling Validation¶
KFold 등과 달리, sampling with replacement (복원추출)에 기반함.
sci-kit learn에서는 sklearn.ensemble.BaggingRegressor 와 sklearn.ensemble.BaggingClassifier로 쉽게 구현할 수 있음.
- 이들은 bagging (Bootstrap + aggregating)을 통한 ensemble model을 만드는 방법임.
- 이를 이용해서 특정 모델의 generalization을 검증하는데에 사용할 수도 있음.
- 단, oob score를 변경할 수 없기 때문에 각 estimator들의 oob samples에 직접 접근하여 성능을 평가하는 루틴을 수동으로 추가해야함.
아래 코드는 Out of Bag을 활용하여 LinearRegressor의 generalization을 Bootstrap Sampling으로 평가함.
import numpy as np
from sklearn.ensemble import BaggingRegressor
from sklearn.datasets import load_diabetes
from sklearn.tree import DecisionTreeRegressor
from sklearn.linear_model import LinearRegression as LinearRegressor
from sklearn.model_selection import train_test_split
from sklearn.metrics import make_scorer
# RMSPE 계산 함수 정의 (0으로 나누는 경우 처리)
def rmspe(y_true, y_pred):
"""
RMSPE 계산 함수 (Root Mean Squared Percentage Error)
:param y_true: 실제 값 (numpy array)
:param y_pred: 예측 값 (numpy array)
:return: RMSPE 값 (% 단위)
y_true가 0인 경우 해당 요소는 0으로 처리하여 나누기 오류를 방지합니다.
"""
# y_true가 0이 아닌 경우만 RMSPE 계산
mask = y_true != 0
rmspe_value = np.sqrt(np.mean(np.where(mask, ((y_true[mask] - y_pred[mask]) / y_true[mask]) ** 2, 0)))
return rmspe_value * 100 # 퍼센트 단위로 반환
# 데이터 로드 (당뇨병 데이터 사용)
diabetes = load_diabetes()
X_train, y_train = diabetes.data, diabetes.target # 특성 및 타겟 값
# BaggingRegressor에서 부트스트래핑 및 OOB 사용 (oob_score=True)
bagging = BaggingRegressor(
estimator=LinearRegressor(), # 기본 모델로 선형 회귀 사용
n_estimators=100, # 더 많은 부트스트랩 샘플 사용 (100개의 부트스트랩 샘플)
bootstrap=True, # 복원 추출(부트스트래핑)
oob_score=True, # OOB 스코어 계산 (훈련에 사용되지 않은 샘플로 평가)
random_state=42 # 재현성을 위한 랜덤 시드
)
# 모델 학습 (부트스트래핑을 통해 100개의 부트스트랩 샘플로 학습)
bagging.fit(X_train, y_train)
# OOB RMSPE 수동 계산을 위한 초기화
tmp = []
oob_sizes = []
# 각 부트스트랩 모델에서 OOB 성능을 계산하기 위해 각 모델에 접근
for i, estimator in enumerate(bagging.estimators_):
# i번째 모델의 OOB 샘플 추출 (훈련에 사용되지 않은 샘플들)
oob_samples = np.setdiff1d(np.arange(X_train.shape[0]), bagging.estimators_samples_[i])
# i번째 모델의 OOB 샘플에 대한 예측 수행
oob_predictions = estimator.predict(X_train[oob_samples])
# i번째 모델의 OOB 샘플에 대한 RMSPE 계산
oob_rmspe = rmspe(y_train[oob_samples], oob_predictions)
# OOB RMSPE 및 샘플 크기 저장
tmp.append(oob_rmspe)
oob_sizes.append(len(oob_samples)) # OOB 샘플의 크기 기록
# 수동으로 계산한 각 모델의 OOB RMSPE 값을 numpy 배열로 변환
oob_rmspes = np.array(tmp)
# 자동으로 제공되는 OOB R² 점수 출력 (전체 모델에 대한 OOB 평가)
print(f"OOB R^2 score (BaggingRegressor): {bagging.oob_score_:.4f}")
# 수동으로 계산된 OOB 샘플 크기 출력
print(f'OOB 샘플 크기: {oob_sizes}')
# 각 부트스트랩 모델의 OOB RMSPE 출력
print(f"OOB RMSPE: {np.round(oob_rmspes, 4)}") # 각 모델의 RMSPE 출력
print(f"Mean RMSPE: {np.mean(oob_rmspes):.4f}") # OOB RMSPE 평균 출력
print(f"Standard deviation of RMSPE: {np.std(oob_rmspes):.4f}") # OOB RMSPE 표준 편차 출력
결과는 다음과 같음:
OOB R^2 score (BaggingRegressor): 0.4939
OOB 샘플 크기: [169, 167, 171, 166, 174, 165, 156, 165, 162, 173, 167, 168, 160, 165, 151, 162, 162, 166, 166, 158, 157, 170, 165, 165, 160, 163, 158, 165, 160, 173, 163, 159, 164, 173, 162, 150, 163, 168, 166, 156, 167, 161, 167, 166, 169, 162, 154, 164, 163, 170, 166, 154, 169, 156, 163, 173, 164, 162, 167, 163, 167, 166, 163, 151, 160, 157, 170, 165, 160, 165, 159, 171, 174, 164, 164, 168, 168, 157, 168, 161, 165, 167, 163, 168, 164, 162, 156, 156, 154, 164, 165, 172, 166, 171, 171, 163, 175, 164, 163, 163]
OOB RMSPE: [61.9002 68.1398 54.0005 79.3851 60.2074 58.9296 63.8427 76.783 52.4628
64.8675 55.598 52.1469 64.3121 55.9179 55.7934 53.3193 73.826 73.1933
62.474 52.7245 68.7434 40.4269 65.1143 55.0646 51.4775 46.614 62.5718
78.5828 49.6883 52.7509 58.2236 69.4002 73.4784 51.3313 65.1581 70.3054
48.5123 65.0442 59.9873 68.0095 58.2426 65.6644 70.1878 57.033 54.8067
72.6428 54.6682 60.3641 68.1186 57.2257 48.9798 59.2695 61.7272 74.8815
65.7823 52.8685 75.8178 51.4954 80.3629 65.414 61.9127 60.0376 58.7429
65.3099 51.2952 52.7607 74.0237 64.7868 53.888 53.5887 67.056 64.2244
58.5203 59.322 67.2109 68.72 50.9752 58.3827 67.6617 58.9911 76.2023
64.517 56.8323 58.6692 55.1377 54.8658 54.1806 71.4863 65.4693 49.6209
57.809 65.3187 52.0799 54.657 52.9604 62.7187 57.4264 50.5707 68.7582
79.058 ]
Mean RMSPE: 61.2161
Standard deviation of RMSPE: 8.4207